「我們看起來都很健康,寶寶應該不會有問題吧?」這是許多準爸媽在備孕前的自然反應。但這句話,可能藏著我們沒發現的基因風險。畢竟,看起來一切正常,怎麼會想到基因裡可能藏著風險?其實,即使夫妻雙方外表健康、無家族病史,也有機會攜帶特定遺傳疾病的「隱性基因」。
根據過去帶因篩檢的研究文獻指出,從傳統(較小)基因檢測套組到中大型套組的研究中,約30%到超過50%的人是至少一項遺傳疾病的帶因者。當伴侶或是夫妻雙方攜帶相同的致病基因,或是女方帶有性聯遺傳致病基因時,就有 ¼ 的機率生下患病的寶寶。該如何防範這種看不見的風險,是預防醫學一項重要的課題。
送子鳥實驗室自 2022 年 4 月推出愛基因(iGene)檢測,以疾病嚴重性、早期發病、亞洲高盛行率等條件,作為挑選基因的依據。只需抽血就能一次篩檢 350 個基因、31784 個變異位點的 DNA 是否正常,並根據雙方的檢測結果進而計算出未來寶寶的罹病風險。截至 2025年 6 月,累計超過 1500 個人進行愛基因檢測。
帶有隱性遺傳疾病基因變異的比例 & 變異基因排名
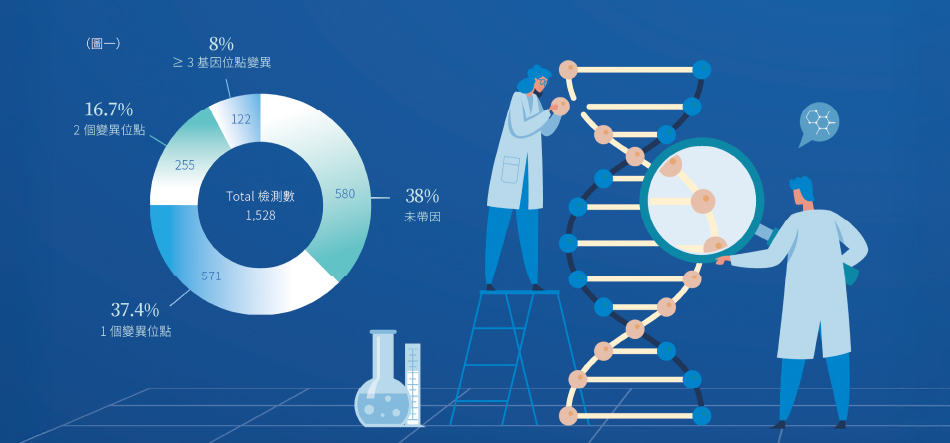
愛基因的檢測結果顯示:38%(580/1528)的檢測者未帶有突變基因位點、37.4%(571/1528)帶有1個突變位點、16.7%(255/1528)帶有2個突變基因位點、8%(122/1528)帶有3個以上的突變基因位點(圖一)。
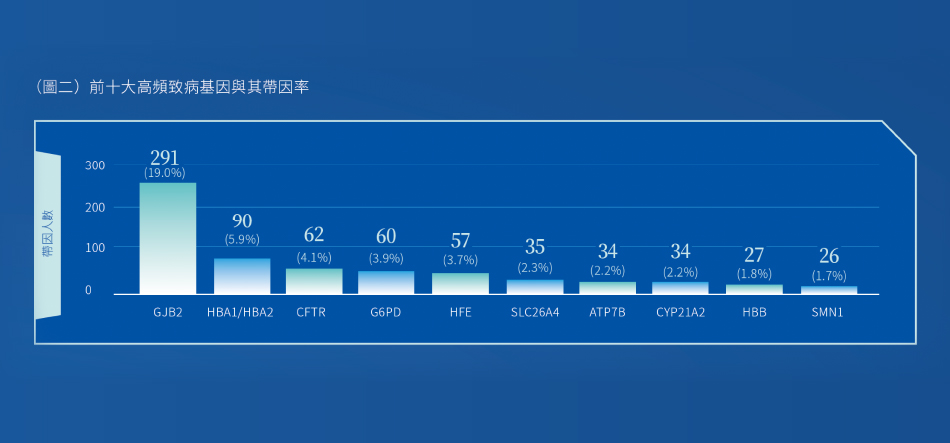
進一步針對 350 個基因的變異出現率進行分析,頻率由高至低排序依序為:GJB2、HBA1/2、CFTR、G6PD、HFE(圖二)。
以過去文獻(Wu et al., 2011)¹ 提到的新生兒同型合子發病率(1.08%)回推帶因率,也差不多是這個數字,其中 95% 的 GJB2 基因變異都是 c.109G>A 這個位點。排名第二的基因 HBA1/2 正是大家耳熟能詳的地中海型貧血,愛基因檢測其帶因率為 5.9%,也與過去台灣文獻的 5~8% 相近(Liao et al., 2023)²。
配對到相同基因變異的比例(At‑Risk Couple)
接下來,讓我們把焦點從「個人檢測」轉向「伴侶、夫妻或卵子捐/受贈者」雙方同時進行檢測的結果。在ECS檢測中,用於評估雙方檢測風險的指標為「同病帶因伴侶」(At‑Risk Couple,ARC),指的是夫妻雙方帶有相同的隱性遺傳疾病基因,或女性是性聯遺傳疾病的帶因者,代表他們的下一代有罹患疾病的風險。
根據愛基因檢測的大數據,在 623 對伴侶、夫妻或卵子捐/受贈者中,有:
- 52 對雙方都帶有相同隱性遺傳疾病的基因變異(占 8.3%)
- 44 位女性為性聯遺傳疾病帶因者
無論是否已經進行愛基因檢測, 都可以透過「愛基因(iGene)遺傳疾病帶因風險計算機」來快速評估風險。風險計算機針對愛基因所涵蓋的 350 個基因,可快速計算出夫妻的潛在罹病風險,大幅節省人工解說與諮詢時間,讓您更快掌握下一步的選擇。
ECS 檢測後的下一步
愛基因檢測完成後,解讀與後續行動同樣關鍵。根據美國多個權威學會( ACMG、ACOG、ASRM)的共識建議,將檢測後結果分三大類,列出後續建議動作:

根據檢測結果採取適當的後續行動,不僅能協助伴侶做出知情的生育選擇,也能有效降低遺傳疾病傳遞給下一代的風險,實現預防醫學的核心目標。
擴展性遺傳帶因篩檢(ECS)早已不只是高風險族群的專利,而是每一對準備迎接新生命伴侶都值得擁有的「孕前檢查新標配」,因為預防,永遠勝過遺憾。愛基因檢測能讓未來父母在懷孕前,就掌握風險來源、了解對策,做出知情且主動的選擇,將風險降到最低。
讓我們透過愛基因檢測,從現在開始為下一代打造一個沒有遺傳病風險的未來。
參考資料:
- Wu, C.-C., Hung, C.-C., Lin, S.-Y., Hsieh, W.-S., Tsao, P.-N., Lee, C.-N., Su, Y.-N., & Hsu, C.-J.(2011, July 19).Newborn genetic screening for hearing impairment: A preliminary study at a tertiary center.PLOS ONE, 6(7), e22314.https://doi.org/10.1371/journal.pone.0022314
- Liao, W.-C., Lee, Y.-P., Chang, C.-Y., Wang, J.-W., Chen, P.-E., & Lin, M.-H.(2023).Complete genomic profiles of 1,496 Taiwanese reveal curated insights into population and individual medical potential.Computational and Structural Biotechnology Journal, 21, 345–362



